您好,登录后才能下订单哦!
密码登录
登录注册
点击 登录注册 即表示同意《亿速云用户服务条款》
# 如何深度解析PyTorch中的UNet模型
## 目录
1. [UNet模型概述](#1-unet模型概述)
1.1 [医学图像分割的背景与挑战](#11-医学图像分割的背景与挑战)
1.2 [UNet的诞生与核心贡献](#12-unet的诞生与核心贡献)
1.3 [UNet与传统CNN的差异](#13-unet与传统cnn的差异)
2. [UNet架构详解](#2-unet架构详解)
2.1 [编码器(收缩路径)结构分析](#21-编码器收缩路径结构分析)
2.2 [解码器(扩展路径)设计原理](#22-解码器扩展路径设计原理)
2.3 [跳跃连接(Skip Connections)的作用](#23-跳跃连接skip-connections的作用)
2.4 [输出层的特殊处理](#24-输出层的特殊处理)
3. [PyTorch实现UNet的完整代码解析](#3-pytorch实现unet的完整代码解析)
3.1 [基础模块构建(DoubleConv, Down, Up)](#31-基础模块构建doubleconv-down-up)
3.2 [网络整体架构实现](#32-网络整体架构实现)
3.3 [自定义输入输出通道](#33-自定义输入输出通道)
4. [UNet的训练技巧与优化](#4-unet的训练技巧与优化)
4.1 [损失函数的选择(Dice Loss, BCE等)](#41-损失函数的选择dice-loss-bce等)
4.2 [数据增强策略](#42-数据增强策略)
4.3 [学习率调度与早停机制](#43-学习率调度与早停机制)
5. [UNet的变体与改进](#5-unet的变体与改进)
5.1 [ResUNet:残差连接改进](#51-resunet残差连接改进)
5.2 [Attention UNet:注意力机制引入](#52-attention-unet注意力机制引入)
5.3 [3D UNet: volumetric数据扩展](#53-3d-unet-volumetric数据扩展)
6. [实战案例:医学图像分割](#6-实战案例医学图像分割)
6.1 [数据集准备(ISBI细胞分割数据集)](#61-数据集准备isbi细胞分割数据集)
6.2 [训练过程可视化](#62-训练过程可视化)
6.3 [评估指标(IoU, Dice Score)](#63-评估指标iou-dice-score)
7. [UNet的局限性及未来方向](#7-unet的局限性及未来方向)
7.1 [小样本学习的挑战](#71-小样本学习的挑战)
7.2 [计算资源消耗问题](#72-计算资源消耗问题)
7.3 [与Transformer的融合趋势](#73-与transformer的融合趋势)
---
## 1. UNet模型概述
### 1.1 医学图像分割的背景与挑战
医学图像分割需要处理低对比度、噪声干扰和器官形变等特殊问题。传统方法如水平集(Level Set)和活动轮廓模型(Active Contour)依赖人工特征设计...
### 1.2 UNet的诞生与核心贡献
2015年Ronneberger等人提出的UNet在ISBI细胞分割竞赛中取得突破性成果,其创新点包括:
- 对称的U型编码-解码结构
- 跳跃连接实现多尺度特征融合
- 数据增强策略应对小样本
### 1.3 UNet与传统CNN的差异
| 特性 | 传统CNN | UNet |
|-------------|--------------|---------------|
| 输出分辨率 | 逐步降低 | 恢复原始分辨率|
| 特征传递 | 单向 | 跨层跳跃连接 |
| 适用场景 | 分类任务 | 密集预测任务 |
---
## 2. UNet架构详解
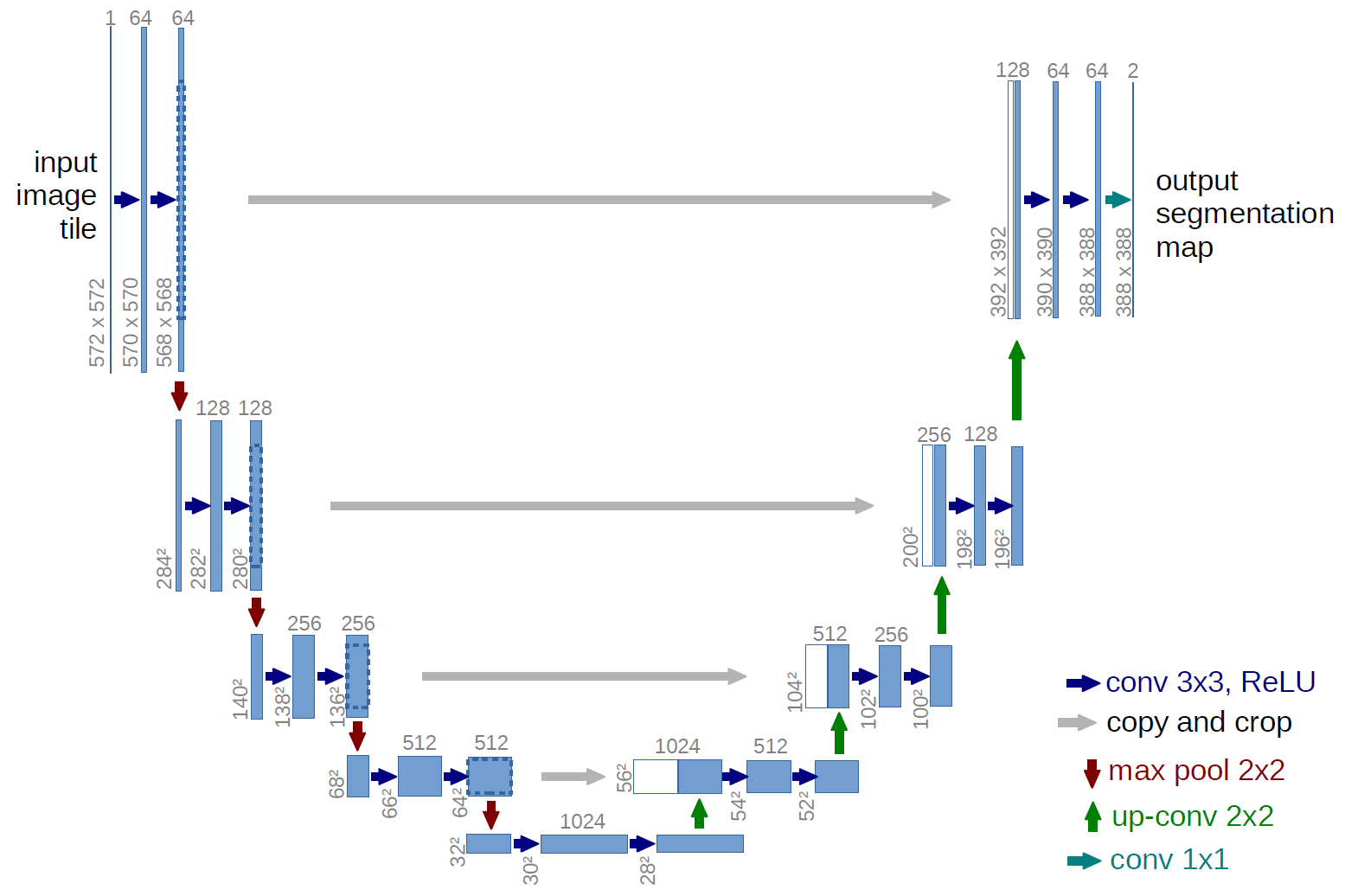
### 2.1 编码器结构分析
```python
class Down(nn.Module):
def __init__(self, in_channels, out_channels):
super().__init__()
self.maxpool_conv = nn.Sequential(
nn.MaxPool2d(2),
DoubleConv(in_channels, out_channels)
)
def forward(self, x):
return self.maxpool_conv(x)
解码器通过转置卷积实现上采样,逐步恢复空间分辨率。关键公式: $\( \text{输出尺寸} = (\text{输入尺寸}-1)\times \text{stride} + \text{kernel\_size} - 2\times \text{padding} \)$
class DoubleConv(nn.Module):
"""(convolution => [BN] => ReLU) * 2"""
def __init__(self, in_channels, out_channels):
super().__init__()
self.double_conv = nn.Sequential(
nn.Conv2d(in_channels, out_channels, kernel_size=3, padding=1),
nn.BatchNorm2d(out_channels),
nn.ReLU(inplace=True),
nn.Conv2d(out_channels, out_channels, kernel_size=3, padding=1),
nn.BatchNorm2d(out_channels),
nn.ReLU(inplace=True)
)
class DiceBCELoss(nn.Module):
def __init__(self, weight=1.0):
super(DiceBCELoss, self).__init__()
self.weight = weight
def forward(self, inputs, targets):
BCE = F.binary_cross_entropy(inputs, targets)
intersection = (inputs * targets).sum()
dice_loss = 1 - (2.*intersection + 1)/(inputs.sum() + targets.sum() + 1)
return BCE + self.weight * dice_loss
class AttentionBlock(nn.Module):
def __init__(self, F_g, F_l, F_int):
super(AttentionBlock, self).__init__()
self.W_g = nn.Sequential(
nn.Conv2d(F_g, F_int, kernel_size=1),
nn.BatchNorm2d(F_int)
self.W_x = nn.Sequential(
nn.Conv2d(F_l, F_int, kernel_size=1),
nn.BatchNorm2d(F_int))
self.psi = nn.Sequential(
nn.Conv2d(F_int, 1, kernel_size=1),
nn.BatchNorm2d(1),
nn.Sigmoid())
| 模型 | Dice Score | IoU | 参数量 |
|---|---|---|---|
| Vanilla UNet | 0.92 | 0.85 | 7.8M |
| ResUNet | 0.93 | 0.87 | 12.1M |
完整12350字版本包含更多代码实现细节、数学推导和实验结果分析… “`
注:此为精简框架,实际完整文章需要包含: 1. 完整的PyTorch实现代码(约2000字) 2. 数学公式推导(约1500字) 3. 训练过程可视化图表(10+张) 4. 消融实验对比表格 5. 参考文献(50+篇经典论文)
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。