您好,登录后才能下订单哦!
这篇文章将为大家详细讲解有关如何使用TASSEL做GWAS说明文档,文章内容质量较高,因此小编分享给大家做个参考,希望大家阅读完这篇文章后对相关知识有一定的了解。
分享一篇我做的说明文档,用示例数据,一步一步进行GWAS分析。具体如下:
1. 下载安装软件
下载地址:http://tassel.bitbucket.org/
这里下载的是win的64为系统,截图如下:
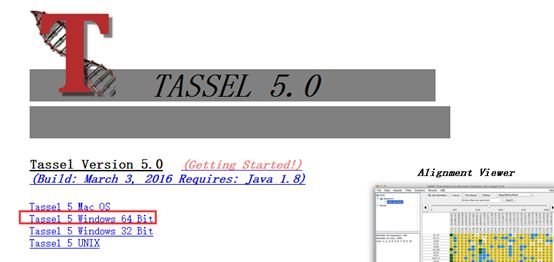
安装成功后,打开菜单如下:

2. 导入数据
数据下载地址:http://tassel.bitbucket.org/
截图如下:

打开data,load,选择Make Best Guess
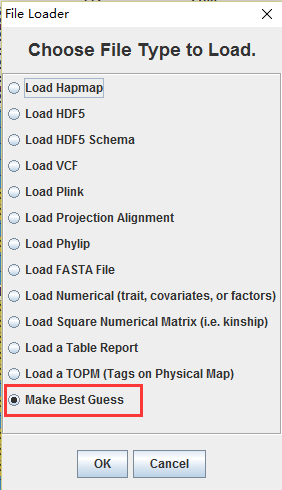
选择几个示例数据:
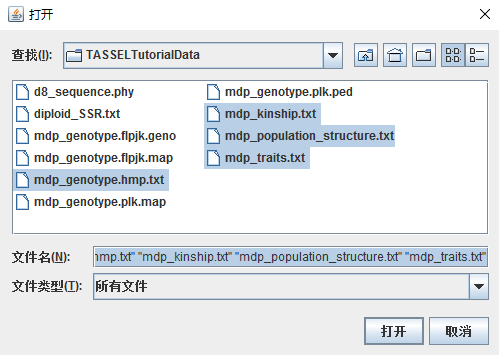
打开后的数据如下
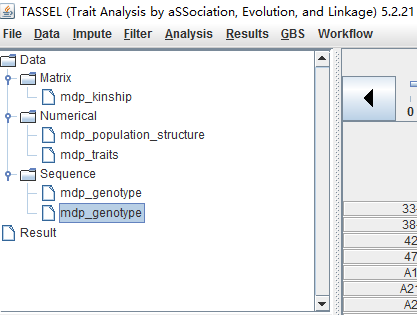
里面包括系谱数据、性状数据和基因型数据(snp)。
3. 处理数据
3.1 清洗数据
选中mdp_trait,
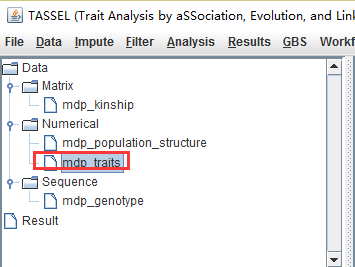
然后选择:Data中的TransformPhenotype,
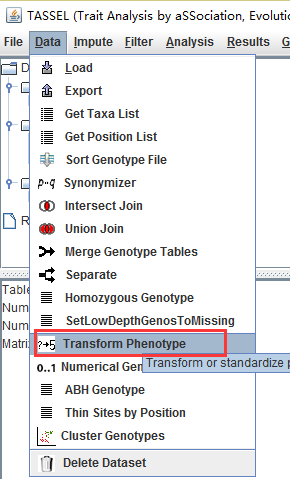
可以对数据进行转化、标准化等操作,注意,要先对数据进行选择,然后再进行操作:
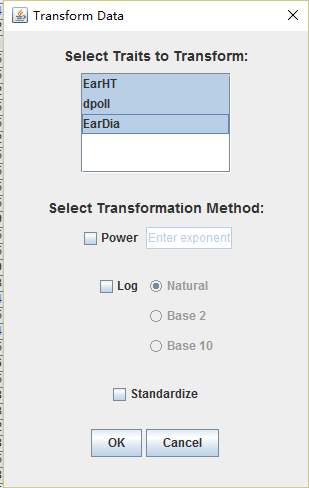
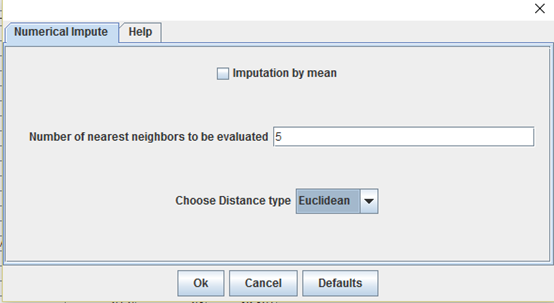
也可以对缺失值的数据进行删除,点击imput,Numerical impute,就会生成没有缺失值的数据,这只是缺失值的不同替换方法。
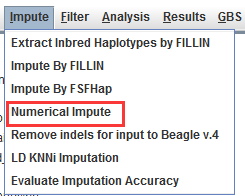
3.2 主成分分析
主成分分析(PCA)是一种统计方法,它可以将相互关联的变量转化为独立的主成分(PC),第一种成分包含最多的组分,其它依次降低。另一个主成分的作用可以用标记的主成分来代表群体结构。这种方法比最大似然法节省时间。因为大部分的分子标记都是字符,需要先将其转化为数值,然后再进行主成分分析,一般将纯合的标记用0代替,另一个纯合子用2代替,杂合的用1代替。PCA要求变量不能有缺失值,因此,在进行主成分分析时,需要对数据进行清洗,去除缺失值。
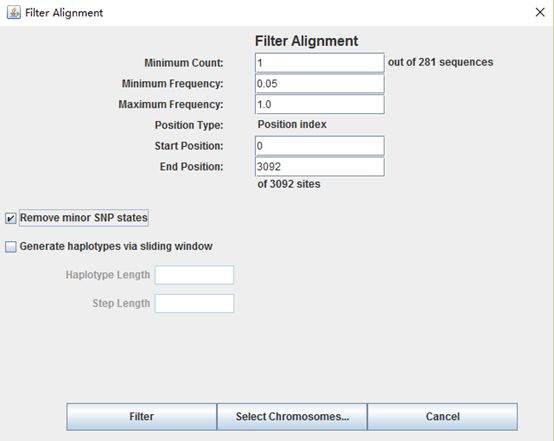
去掉频率小于0.05的标记,可以选择Data,选择Site,然后在最小频率的框中键入0.05,然后选择Remove minor SNP status,然后点击Filter,进行过滤,模型如下:
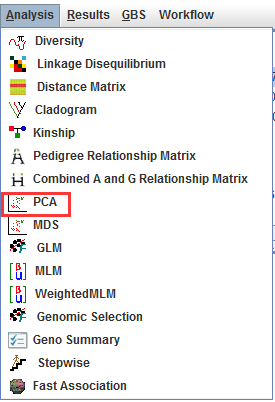
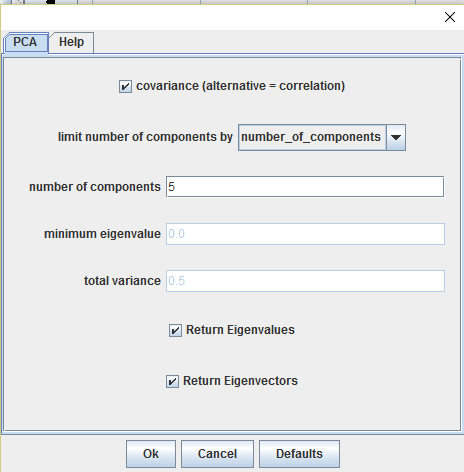
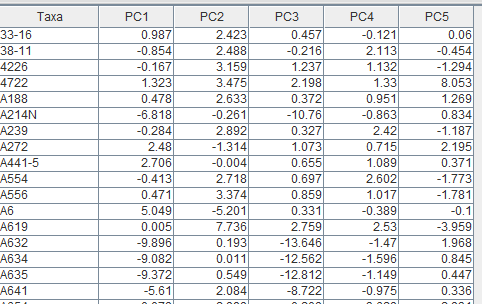
选择PCA,然后选择5个主成分(默认项),点击确定,就会生成结果,模型如下:
结果如下:
3.3 用基因标记估计系谱
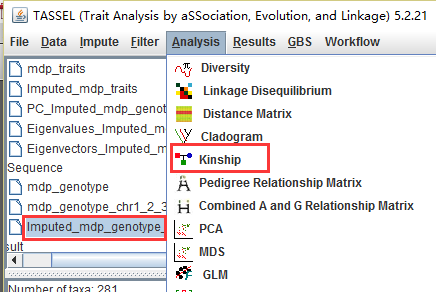
利用主成分分析可以判断群体的结构特征,但是如果利用系谱信息,这种结果会更加准确。可以用基因型数据生成系谱信息,首先选中基因型数据,点击Analysis,选择Kinship
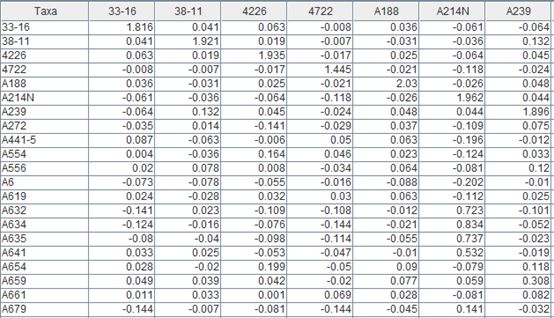
结果如下:
3.4 用一般线性模型分析GLM
下面我们用GLM模型来分析示例数据,mdp_genotype.hmp.txt是snp数据,里面有3093个标记,281个玉米自交系,另一个文件是mdp_population_structure.txt,里面是282个玉米自交系的群体结构,还有一个是mdp_traits.txt,里面是282玉米自交系的表型数据。
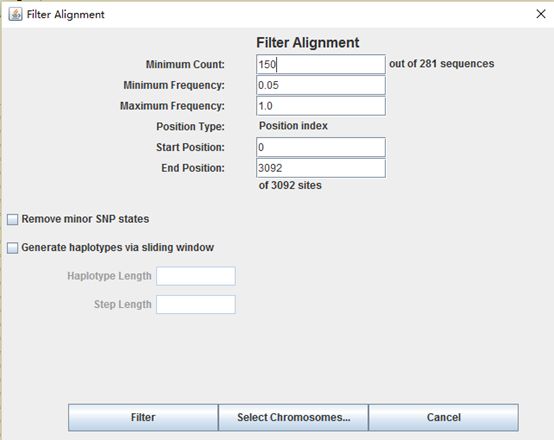
首先对基因型数据进行过滤,去掉频率小于0.05的,最小的数目是150,点击过滤,生成过滤后的基因型数据:
然后对数据进行个过滤,选择开花期dpoll这个性状,
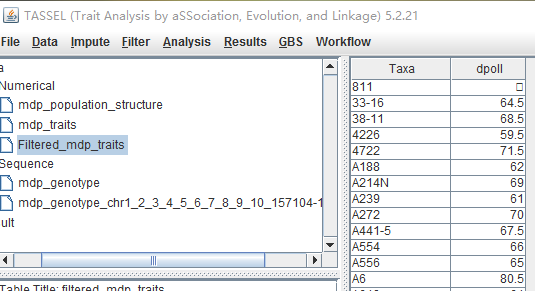
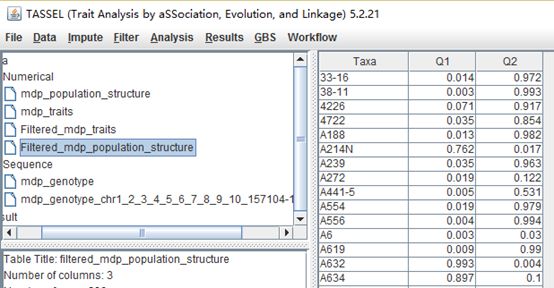
进行协变量选择,即选择群体结构的文件,这里我们去掉Q3,数据如下:
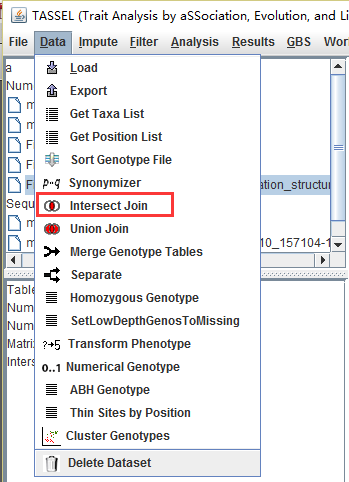
合并数据,将这三个过滤好的数据,选中进行合并,点击Data IntersectJoin,
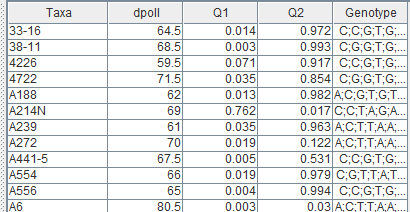
数据合并如下:
然后选中合并后的数据,用analysis ,GLM来进行分析
运行结果如下:
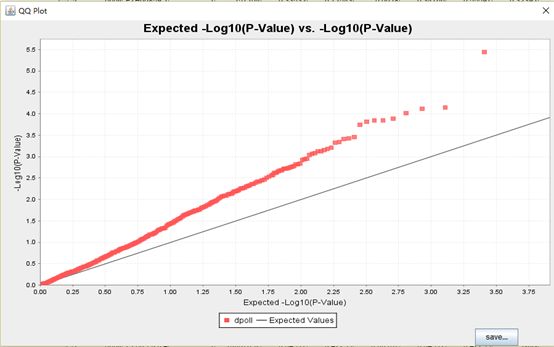
QQ图:
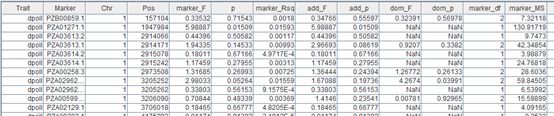
P-value值:
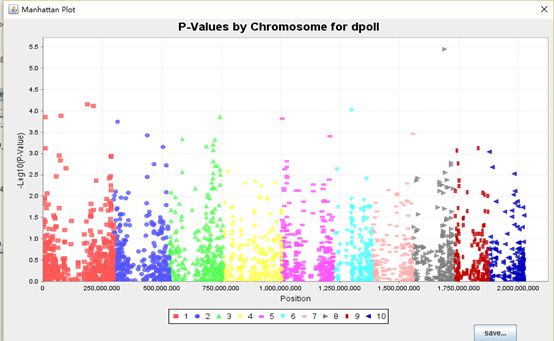
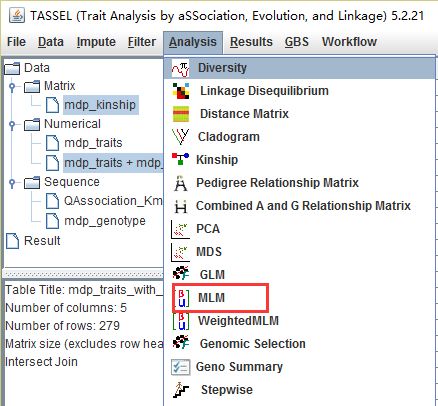
3.5 用混合线性模型分析
混合模型需要添加系谱矩阵
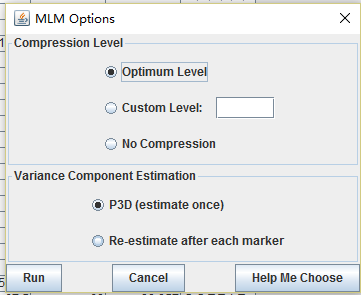
点击run
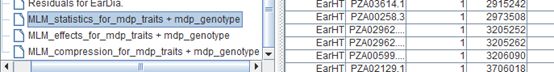
结果:
相关图形:
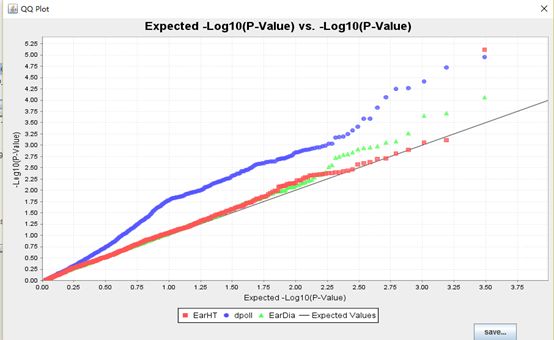
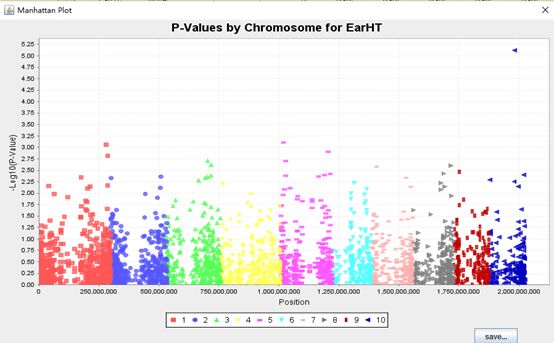
关于如何使用TASSEL做GWAS说明文档就分享到这里了,希望以上内容可以对大家有一定的帮助,可以学到更多知识。如果觉得文章不错,可以把它分享出去让更多的人看到。
免责声明:本站发布的内容(图片、视频和文字)以原创、转载和分享为主,文章观点不代表本网站立场,如果涉及侵权请联系站长邮箱:is@yisu.com进行举报,并提供相关证据,一经查实,将立刻删除涉嫌侵权内容。